Post-translationale Proteinmodifikationen
Neben der Lokalisierung der durch post-translationale Prozesse modifizierten Aminosäuren, erlaubt die NMR-Spektroskopie zusätzlich den Einfluss auf die Struktur und die Wechselwirkungen des modifizierten Proteins zu bestimmen. In diesem Zusammenhang konnten wir die Regulation des CDK-Inhibitors p19INK4d im humanen Zellzyklus durch Phosphorylierung (Abbildung) aufklären.
Auch während dem Alterungsprozess von Proteinen kommt es zu Modifizierungen, die zu Krankheiten wie z.B. dem grauen Star führen können. Hierzu untersuchen wir den Einfluss von UV-Strahlung und der Glykierung auf Proteine der Augenlinse.
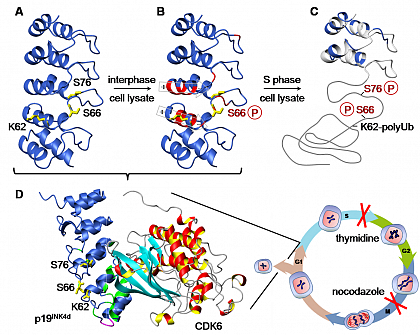
Regulation des CDK-Inhibitors p19INK4d im humanen Zellzyklus durch Phosphorylierung.

