Strukturbestimmung von Proteinen
Mit Hilfe der NMR-Spektroskopie ist es möglich, die hochaufgelöste dreidimensionale Struktur von Biomakromolekülen zu bestimmen. Hierzu werden mehrere tausend Atomabstände auf 0.2-0.5 nm genau bestimmt. Weiter fließen eine Vielzahl von Bindungswinkel und die relative Orientierungen von chemischen Bindungen ein. Aus diesen experimentellen Daten wird die 3D-Struktur über eine Moleküldynamikrechnung bestimmt.
Für folgende Proteine konnte die Struktur mittels NMR-Spektroskopie bestimmt werden: Kälteschockprotein CspB aus Bacillus subtilis (2F52.pdb), Parvulindomäne PpiD aus E. coli (2KGJ.pdb), plasmid copy control protein ORF56 aus Sulfolobus islandicus (2K9I.pdb), Prolylisomerase und molekulares Chaperons SlyD (2K8I.pdb) und SlpA (2M2A.pdb), Helix-0 der humanen N-BAR Domäne in SDS (2RMY.pdb) und DPC Mizellen (2RND.pdb), Auxin Transkriptionsfaktor IAA4 aus Pisum Sativum (2M1M.pdb).
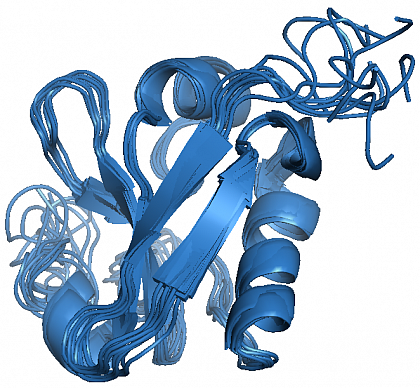
NMR-Struktur von PsIAA4 (Ensemble der 9 Strukturen niedrigster Energie).
Über Kooperationen konnten die Strukturen folgender Proteine mittels Röntgenkristallographie bestimmt werden: Kälteschockproteine sowie deren Komplexe mit einzelsträngigen Nukleinsäuren (2ES2.pdb, 2HAX.pdb, 3PF4.pdb, 3PF5.pdb), ankyrin repeat protein tANK aus Thermoplasma volcanium (2RFM.pdb), Prolylisomerase und molekulares Chaperon SlyD aus Thermus thermophilus (3CGM.pdb, 3CGN.pdb, 3LUO.pdb), plasmid copy control protein ORF56 aus Sulfolobus islandicus (3FT7.pdb), zinc-finger containing Kti11 aus Saccharomcyes cerevisiae (5AX2.pdb), Onconase C87A/C104A aus Rana pipiens (3HG6.pdb), Pilus und TolA Bindedomänen der IF1 und IKe Phagen (4EO0.pdb, 4EO1.pdb, 2X9A.pdb, 2X9B.pdb, 3KNQ.pdb).
Strukturuntersuchen mittels Kryo-Elektronenmikroskopie des humanen Proteins Bin1 N-BAR, welches an der Krümmung von Membranen beteiligt ist, gelangen ebenfalls über Kooperationen.

